 Primeira página > Programação > Dominando a segmentação de imagens: como as técnicas tradicionais ainda brilham na era digital
Primeira página > Programação > Dominando a segmentação de imagens: como as técnicas tradicionais ainda brilham na era digital
Dominando a segmentação de imagens: como as técnicas tradicionais ainda brilham na era digital
Introdução
A segmentação de imagens, um dos procedimentos mais básicos em visão computacional, permite que um sistema decomponha e analise várias regiões dentro de uma imagem. Quer você esteja lidando com reconhecimento de objetos, imagens médicas ou direção autônoma, a segmentação é o que divide as imagens em partes significativas.
Embora os modelos de aprendizagem profunda continuem a ser cada vez mais populares nesta tarefa, as técnicas tradicionais de processamento digital de imagens ainda são poderosas e práticas. As abordagens revisadas nesta postagem incluem limiar, detecção de borda, baseada em região e agrupamento, implementando um conjunto de dados bem reconhecido para a análise de imagens de células, o conjunto de dados de imagens MIVIA HEp-2.
Conjunto de dados de imagens MIVIA HEp-2
O conjunto de dados de imagens MIVIA HEp-2 é um conjunto de imagens de células usadas para analisar o padrão de anticorpos antinucleares (ANA) através de células HEp-2. Consiste em fotos 2D tiradas por microscopia de fluorescência. Isso o torna muito adequado para tarefas de segmentação, principalmente aquelas relacionadas à análise de imagens médicas, onde a detecção de regiões celulares é mais importante.
Agora, vamos passar para as técnicas de segmentação usadas para processar essas imagens, comparando seu desempenho com base nas pontuações da F1.
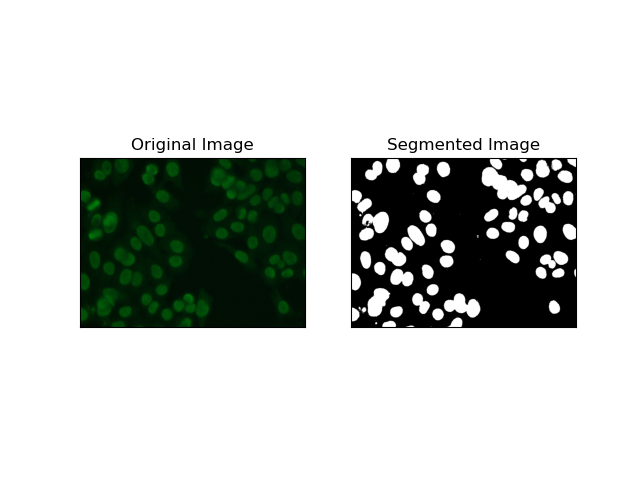
1. Segmentação de limite
Limiar é o processo pelo qual imagens em tons de cinza são convertidas em imagens binárias com base nas intensidades de pixel. No conjunto de dados MIVIA HEp-2, este processo é útil na extração de células do fundo. É simples e eficaz em um nível relativamente grande, especialmente com o método de Otsu, pois ele auto-calcula o limite ideal.
Método de Otsu é um método de limiarização automática, onde tenta encontrar o melhor valor de limiar para produzir a variância intraclasse mínima, separando assim as duas classes: primeiro plano (células) e fundo. O método examina o histograma da imagem e calcula o limite perfeito, onde a soma das variações de intensidade de pixel em cada classe é minimizada.
# Thresholding Segmentation
def thresholding(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Otsu's thresholding
_, thresh = cv.threshold(gray, 0, 255, cv.THRESH_BINARY cv.THRESH_OTSU)
return thresh
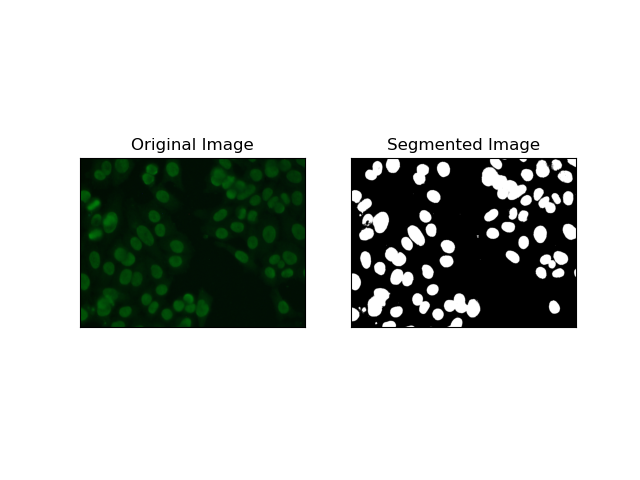
2. Segmentação de detecção de borda
A detecção de bordas refere-se à identificação de limites de objetos ou regiões, como bordas de células no conjunto de dados MIVIA HEp-2. Dos muitos métodos disponíveis para detectar mudanças abruptas de intensidade, o Canny Edge Detector é o melhor e, portanto, o método mais apropriado para ser usado para detectar limites celulares.
Canny Edge Detector é um algoritmo de vários estágios que pode detectar as bordas detectando áreas de fortes gradientes de intensidade. O processo incorpora suavização com filtro gaussiano, cálculo de gradientes de intensidade, aplicação de supressão não máxima para eliminar respostas espúrias e uma operação final de limiar duplo para a retenção apenas de bordas salientes.
# Edge Detection Segmentation
def edge_detection(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Gaussian blur
gray = cv.GaussianBlur(gray, (3, 3), 0)
# Calculate lower and upper thresholds for Canny edge detection
sigma = 0.33
v = np.median(gray)
lower = int(max(0, (1.0 - sigma) * v))
upper = int(min(255, (1.0 sigma) * v))
# Apply Canny edge detection
edges = cv.Canny(gray, lower, upper)
# Dilate the edges to fill gaps
kernel = np.ones((5, 5), np.uint8)
dilated_edges = cv.dilate(edges, kernel, iterations=2)
# Clean the edges using morphological opening
cleaned_edges = cv.morphologyEx(dilated_edges, cv.MORPH_OPEN, kernel, iterations=1)
# Find connected components and filter out small components
num_labels, labels, stats, _ = cv.connectedComponentsWithStats(
cleaned_edges, connectivity=8
)
min_size = 500
filtered_mask = np.zeros_like(cleaned_edges)
for i in range(1, num_labels):
if stats[i, cv.CC_STAT_AREA] >= min_size:
filtered_mask[labels == i] = 255
# Find contours of the filtered mask
contours, _ = cv.findContours(
filtered_mask, cv.RETR_EXTERNAL, cv.CHAIN_APPROX_SIMPLE
)
# Create a filled mask using the contours
filled_mask = np.zeros_like(gray)
cv.drawContours(filled_mask, contours, -1, (255), thickness=cv.FILLED)
# Perform morphological closing to fill holes
final_filled_image = cv.morphologyEx(
filled_mask, cv.MORPH_CLOSE, kernel, iterations=2
)
# Dilate the final filled image to smooth the edges
final_filled_image = cv.dilate(final_filled_image, kernel, iterations=1)
return final_filled_image
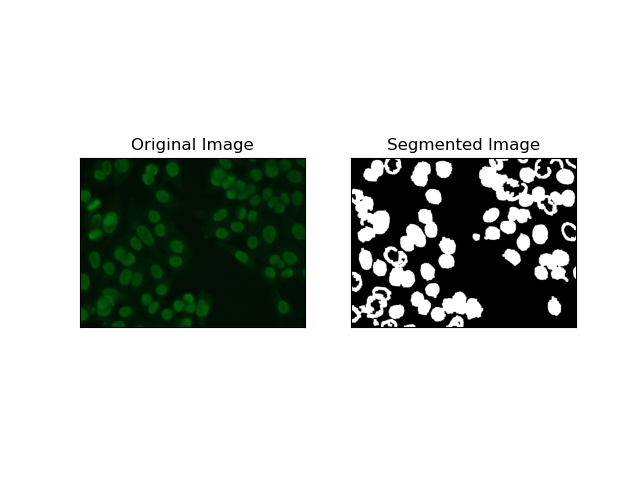
3. Segmentação Baseada em Região
A segmentação baseada em região agrupa pixels semelhantes em regiões, dependendo de determinados critérios, como intensidade ou cor. A técnica
Segmentação de bacias hidrográficas pode ser usada para ajudar na segmentação de imagens de células HEp-2 para poder detectar as regiões que representam células; considera as intensidades dos pixels como uma superfície topográfica e delineia regiões distintas.
Segmentação de bacias hidrográficas trata as intensidades dos pixels como uma superfície topográfica. O algoritmo identifica "bacias" nas quais identifica mínimos locais e depois inunda gradualmente essas bacias para ampliar regiões distintas. Esta técnica é bastante útil quando se deseja separar objetos tocantes, como no caso de células em imagens microscópicas, mas pode ser sensível ao ruído. O processo pode ser guiado por marcadores e a segmentação excessiva muitas vezes pode ser reduzida.
# Edge Detection Segmentation
def edge_detection(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Gaussian blur
gray = cv.GaussianBlur(gray, (3, 3), 0)
# Calculate lower and upper thresholds for Canny edge detection
sigma = 0.33
v = np.median(gray)
lower = int(max(0, (1.0 - sigma) * v))
upper = int(min(255, (1.0 sigma) * v))
# Apply Canny edge detection
edges = cv.Canny(gray, lower, upper)
# Dilate the edges to fill gaps
kernel = np.ones((5, 5), np.uint8)
dilated_edges = cv.dilate(edges, kernel, iterations=2)
# Clean the edges using morphological opening
cleaned_edges = cv.morphologyEx(dilated_edges, cv.MORPH_OPEN, kernel, iterations=1)
# Find connected components and filter out small components
num_labels, labels, stats, _ = cv.connectedComponentsWithStats(
cleaned_edges, connectivity=8
)
min_size = 500
filtered_mask = np.zeros_like(cleaned_edges)
for i in range(1, num_labels):
if stats[i, cv.CC_STAT_AREA] >= min_size:
filtered_mask[labels == i] = 255
# Find contours of the filtered mask
contours, _ = cv.findContours(
filtered_mask, cv.RETR_EXTERNAL, cv.CHAIN_APPROX_SIMPLE
)
# Create a filled mask using the contours
filled_mask = np.zeros_like(gray)
cv.drawContours(filled_mask, contours, -1, (255), thickness=cv.FILLED)
# Perform morphological closing to fill holes
final_filled_image = cv.morphologyEx(
filled_mask, cv.MORPH_CLOSE, kernel, iterations=2
)
# Dilate the final filled image to smooth the edges
final_filled_image = cv.dilate(final_filled_image, kernel, iterations=1)
return final_filled_image
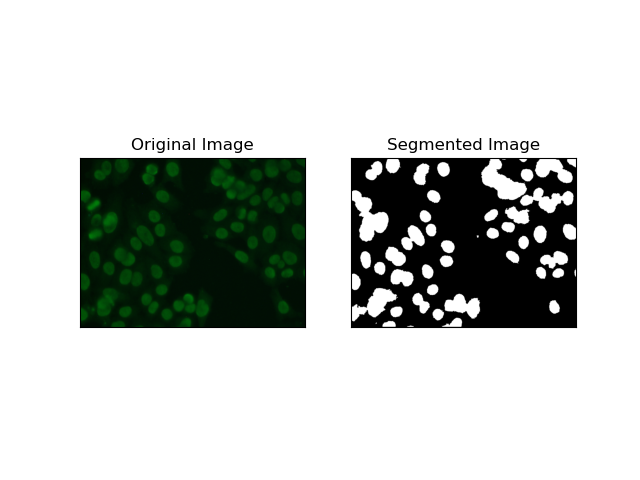
4. Segmentação baseada em cluster
Técnicas de agrupamento como
K-Means tendem a agrupar os pixels em clusters semelhantes, o que funciona bem quando se deseja segmentar células em ambientes multicoloridos ou complexos, como visto em imagens de células HEp-2. Fundamentalmente, isso poderia representar diferentes classes, como uma região celular versus um plano de fundo.
K-means é um algoritmo de aprendizado não supervisionado para agrupar imagens com base na similaridade de pixel de cor ou intensidade. O algoritmo seleciona aleatoriamente K centróides, atribui cada pixel ao centróide mais próximo e atualiza o centróide iterativamente até convergir. É particularmente eficaz na segmentação de uma imagem que possui múltiplas regiões de interesse muito diferentes umas das outras.
# Edge Detection Segmentation
def edge_detection(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Gaussian blur
gray = cv.GaussianBlur(gray, (3, 3), 0)
# Calculate lower and upper thresholds for Canny edge detection
sigma = 0.33
v = np.median(gray)
lower = int(max(0, (1.0 - sigma) * v))
upper = int(min(255, (1.0 sigma) * v))
# Apply Canny edge detection
edges = cv.Canny(gray, lower, upper)
# Dilate the edges to fill gaps
kernel = np.ones((5, 5), np.uint8)
dilated_edges = cv.dilate(edges, kernel, iterations=2)
# Clean the edges using morphological opening
cleaned_edges = cv.morphologyEx(dilated_edges, cv.MORPH_OPEN, kernel, iterations=1)
# Find connected components and filter out small components
num_labels, labels, stats, _ = cv.connectedComponentsWithStats(
cleaned_edges, connectivity=8
)
min_size = 500
filtered_mask = np.zeros_like(cleaned_edges)
for i in range(1, num_labels):
if stats[i, cv.CC_STAT_AREA] >= min_size:
filtered_mask[labels == i] = 255
# Find contours of the filtered mask
contours, _ = cv.findContours(
filtered_mask, cv.RETR_EXTERNAL, cv.CHAIN_APPROX_SIMPLE
)
# Create a filled mask using the contours
filled_mask = np.zeros_like(gray)
cv.drawContours(filled_mask, contours, -1, (255), thickness=cv.FILLED)
# Perform morphological closing to fill holes
final_filled_image = cv.morphologyEx(
filled_mask, cv.MORPH_CLOSE, kernel, iterations=2
)
# Dilate the final filled image to smooth the edges
final_filled_image = cv.dilate(final_filled_image, kernel, iterations=1)
return final_filled_image
Avaliando as técnicas usando pontuações F1
A
pontuação F1 é uma medida que combina precisão e recall para comparar a imagem de segmentação prevista com a imagem real. É a média harmônica de precisão e recuperação, útil em casos de alto desequilíbrio de dados, como em conjuntos de dados de imagens médicas.
Calculamos a pontuação F1 para cada método de segmentação nivelando a verdade básica e a imagem segmentada e calculando a pontuação F1 ponderada.
# Edge Detection Segmentation
def edge_detection(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Gaussian blur
gray = cv.GaussianBlur(gray, (3, 3), 0)
# Calculate lower and upper thresholds for Canny edge detection
sigma = 0.33
v = np.median(gray)
lower = int(max(0, (1.0 - sigma) * v))
upper = int(min(255, (1.0 sigma) * v))
# Apply Canny edge detection
edges = cv.Canny(gray, lower, upper)
# Dilate the edges to fill gaps
kernel = np.ones((5, 5), np.uint8)
dilated_edges = cv.dilate(edges, kernel, iterations=2)
# Clean the edges using morphological opening
cleaned_edges = cv.morphologyEx(dilated_edges, cv.MORPH_OPEN, kernel, iterations=1)
# Find connected components and filter out small components
num_labels, labels, stats, _ = cv.connectedComponentsWithStats(
cleaned_edges, connectivity=8
)
min_size = 500
filtered_mask = np.zeros_like(cleaned_edges)
for i in range(1, num_labels):
if stats[i, cv.CC_STAT_AREA] >= min_size:
filtered_mask[labels == i] = 255
# Find contours of the filtered mask
contours, _ = cv.findContours(
filtered_mask, cv.RETR_EXTERNAL, cv.CHAIN_APPROX_SIMPLE
)
# Create a filled mask using the contours
filled_mask = np.zeros_like(gray)
cv.drawContours(filled_mask, contours, -1, (255), thickness=cv.FILLED)
# Perform morphological closing to fill holes
final_filled_image = cv.morphologyEx(
filled_mask, cv.MORPH_CLOSE, kernel, iterations=2
)
# Dilate the final filled image to smooth the edges
final_filled_image = cv.dilate(final_filled_image, kernel, iterations=1)
return final_filled_image
Em seguida, visualizamos as pontuações F1 de diferentes métodos usando um gráfico de barras simples:
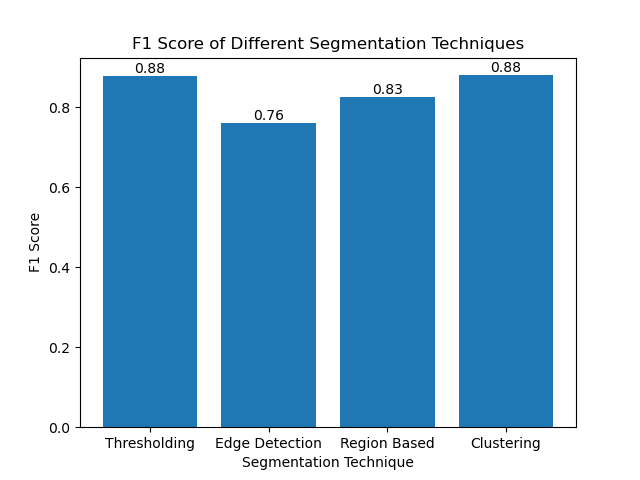
Conclusão
Embora muitas abordagens recentes para segmentação de imagens estejam surgindo, técnicas tradicionais de segmentação, como limiar, detecção de bordas, métodos baseados em região e agrupamento, podem ser muito úteis quando aplicadas a conjuntos de dados como o conjunto de dados de imagens MIVIA HEp-2.
Cada método tem sua força:
- Limiar é bom para segmentação binária simples.
- Edge Detection é uma técnica ideal para a detecção de limites. A segmentação
- baseada em região é muito útil para separar componentes conectados de seus vizinhos. Os métodos de
- Clustering são adequados para tarefas de segmentação multirregional.
Obrigado pela leitura! Espero que esta exploração das técnicas tradicionais de segmentação de imagens inspire seu próximo projeto. Sinta-se à vontade para compartilhar suas idéias e experiências nos comentários abaixo!
-
 Qual método é mais eficiente para a detecção de ponto em polígono: rastreamento de raio ou path.contains_points?detecção de ponto-em-polígono eficiente em python determinar se um ponto está dentro de um polígono é uma tarefa frequente na geometria computac...Programação Postado em 2025-02-19
Qual método é mais eficiente para a detecção de ponto em polígono: rastreamento de raio ou path.contains_points?detecção de ponto-em-polígono eficiente em python determinar se um ponto está dentro de um polígono é uma tarefa frequente na geometria computac...Programação Postado em 2025-02-19 -
 Como definir dinamicamente as teclas em objetos JavaScript?como criar uma chave dinâmica para uma variável de objeto JavaScript ao tentar criar uma chave dinâmica para um objeto JavaScript, usando esta s...Programação Postado em 2025-02-19
Como definir dinamicamente as teclas em objetos JavaScript?como criar uma chave dinâmica para uma variável de objeto JavaScript ao tentar criar uma chave dinâmica para um objeto JavaScript, usando esta s...Programação Postado em 2025-02-19 -
 Como posso verificar com segurança a existência da coluna em uma tabela MySQL?] Outros sistemas de banco de dados. O método comumente tentado: se existe (selecione * de informação_schema.columns Onde tabela_name = ...Programação Postado em 2025-02-19
Como posso verificar com segurança a existência da coluna em uma tabela MySQL?] Outros sistemas de banco de dados. O método comumente tentado: se existe (selecione * de informação_schema.columns Onde tabela_name = ...Programação Postado em 2025-02-19 -
 Como classificar os dados por comprimento da string em mysql usando char_length ()?selecionando dados por comprimento da string em mysql para classificar os dados com base no comprimento da string no mysql, em vez de usar str...Programação Postado em 2025-02-19
Como classificar os dados por comprimento da string em mysql usando char_length ()?selecionando dados por comprimento da string em mysql para classificar os dados com base no comprimento da string no mysql, em vez de usar str...Programação Postado em 2025-02-19 -
 Como verificar se um objeto tem um atributo específico no Python?Método para determinar o atributo de objeto Existence Esta consulta busca um método para verificar a presença de um atributo específico em um ...Programação Postado em 2025-02-19
Como verificar se um objeto tem um atributo específico no Python?Método para determinar o atributo de objeto Existence Esta consulta busca um método para verificar a presença de um atributo específico em um ...Programação Postado em 2025-02-19 -
 Por que não é um pedido de solicitação de captura de entrada no PHP, apesar do código válido?abordando o mau funcionamento da solicitação de postagem em php no snippet de código apresentado: action='' Considere a alocação de memóri...Programação Postado em 2025-02-19
Por que não é um pedido de solicitação de captura de entrada no PHP, apesar do código válido?abordando o mau funcionamento da solicitação de postagem em php no snippet de código apresentado: action='' Considere a alocação de memóri...Programação Postado em 2025-02-19 -
 Como garantir o Hibernate preserva os valores de enum ao mapear para uma coluna MySQL Enum?preservando os valores da enum no hibernate: solucionando problemas de coluna Tipo de coluna no reino da persistência de dados, garantindo a c...Programação Postado em 2025-02-19
Como garantir o Hibernate preserva os valores de enum ao mapear para uma coluna MySQL Enum?preservando os valores da enum no hibernate: solucionando problemas de coluna Tipo de coluna no reino da persistência de dados, garantindo a c...Programação Postado em 2025-02-19 -
 Como resolver discrepâncias do caminho do módulo no Go Mod usando a diretiva substituição?superando a discrepância do caminho do módulo em Go Mod ao utilizar Go Mod, é possível encontrar um conflito em que um pacote de terceiros imp...Programação Postado em 2025-02-19
Como resolver discrepâncias do caminho do módulo no Go Mod usando a diretiva substituição?superando a discrepância do caminho do módulo em Go Mod ao utilizar Go Mod, é possível encontrar um conflito em que um pacote de terceiros imp...Programação Postado em 2025-02-19 -
 Como posso recuperar com eficiência valores de atributo de arquivos XML usando PHP?recuperando valores do atributo dos arquivos xml em php todo desenvolvedor encontra a necessidade de analisar arquivos xml e extrair valores e...Programação Postado em 2025-02-19
Como posso recuperar com eficiência valores de atributo de arquivos XML usando PHP?recuperando valores do atributo dos arquivos xml em php todo desenvolvedor encontra a necessidade de analisar arquivos xml e extrair valores e...Programação Postado em 2025-02-19 -
 O Java permite vários tipos de retorno: uma olhada mais próxima dos métodos genéricos?tipos de retorno múltiplos em java: um equívoco revelado no reino da programação Java, uma assinatura do método peculiar pode surgir, deixando...Programação Postado em 2025-02-19
O Java permite vários tipos de retorno: uma olhada mais próxima dos métodos genéricos?tipos de retorno múltiplos em java: um equívoco revelado no reino da programação Java, uma assinatura do método peculiar pode surgir, deixando...Programação Postado em 2025-02-19 -
 VariedadeOs métodos são FNs que podem ser chamados em objetos Matrizes são objetos, portanto, eles também têm métodos no JS. Flice (Begin): Extra...Programação Postado em 2025-02-19
VariedadeOs métodos são FNs que podem ser chamados em objetos Matrizes são objetos, portanto, eles também têm métodos no JS. Flice (Begin): Extra...Programação Postado em 2025-02-19 -
 Como posso instalar o MySQL no Ubuntu sem um prompt de senha?instalação não interativa de mysql no ubuntu o método padrão de instalar o MySQL Server no Ubuntu usando sudo apt-get install mysql para solic...Programação Postado em 2025-02-19
Como posso instalar o MySQL no Ubuntu sem um prompt de senha?instalação não interativa de mysql no ubuntu o método padrão de instalar o MySQL Server no Ubuntu usando sudo apt-get install mysql para solic...Programação Postado em 2025-02-19 -
 Como posso controlar as vibrações do dispositivo Android com frequências variadas?controlando as vibrações do dispositivo Android com variações de frequência deseja adicionar um elemento tátil ao seu aplicativo Android? Compre...Programação Postado em 2025-02-19
Como posso controlar as vibrações do dispositivo Android com frequências variadas?controlando as vibrações do dispositivo Android com variações de frequência deseja adicionar um elemento tátil ao seu aplicativo Android? Compre...Programação Postado em 2025-02-19 -
 Como superar as restrições de redefinição da função do PHP?superando a função do PHP Redefinição limitações em php, definir uma função com o mesmo nome várias vezes é um não-no. Tentar fazê -lo, como v...Programação Postado em 2025-02-19
Como superar as restrições de redefinição da função do PHP?superando a função do PHP Redefinição limitações em php, definir uma função com o mesmo nome várias vezes é um não-no. Tentar fazê -lo, como v...Programação Postado em 2025-02-19 -
 Quais foram as restrições ao usar o current_timestamp com colunas de registro de data e hora em MySQL antes da versão 5.6.5?restrições em colunas de timestamp com current_timestamp no padrão ou na atualização de cláusulas nas versões MySQL antes de 5.6.5 historicament...Programação Postado em 2025-02-19
Quais foram as restrições ao usar o current_timestamp com colunas de registro de data e hora em MySQL antes da versão 5.6.5?restrições em colunas de timestamp com current_timestamp no padrão ou na atualização de cláusulas nas versões MySQL antes de 5.6.5 historicament...Programação Postado em 2025-02-19
Estude chinês
- 1 Como se diz “andar” em chinês? 走路 Pronúncia chinesa, 走路 aprendizagem chinesa
- 2 Como se diz “pegar um avião” em chinês? 坐飞机 Pronúncia chinesa, 坐飞机 aprendizagem chinesa
- 3 Como se diz “pegar um trem” em chinês? 坐火车 Pronúncia chinesa, 坐火车 aprendizagem chinesa
- 4 Como se diz “pegar um ônibus” em chinês? 坐车 Pronúncia chinesa, 坐车 aprendizagem chinesa
- 5 Como se diz dirigir em chinês? 开车 Pronúncia chinesa, 开车 aprendizagem chinesa
- 6 Como se diz nadar em chinês? 游泳 Pronúncia chinesa, 游泳 aprendizagem chinesa
- 7 Como se diz andar de bicicleta em chinês? 骑自行车 Pronúncia chinesa, 骑自行车 aprendizagem chinesa
- 8 Como você diz olá em chinês? 你好Pronúncia chinesa, 你好Aprendizagem chinesa
- 9 Como você agradece em chinês? 谢谢Pronúncia chinesa, 谢谢Aprendizagem chinesa
- 10 How to say goodbye in Chinese? 再见Chinese pronunciation, 再见Chinese learning
























